参考基因组是一个Fasta格式的文件,里面只有单纯的序列信息。
注释文件即对参考基因组里的序列进行详细注释的文本,常用的有BED、GTF、GFF格式。
常用的基因组及注释文件下载途径有四个:
所有的数据都提供了两种下载方式,一种为网页界面下载,另一种为FTP下载。推荐使用FTP下载。
NCBI
从NCBI下载参考基因组和注释信息:
输入目标物种名(常用名或者学术拉丁名一般都可)
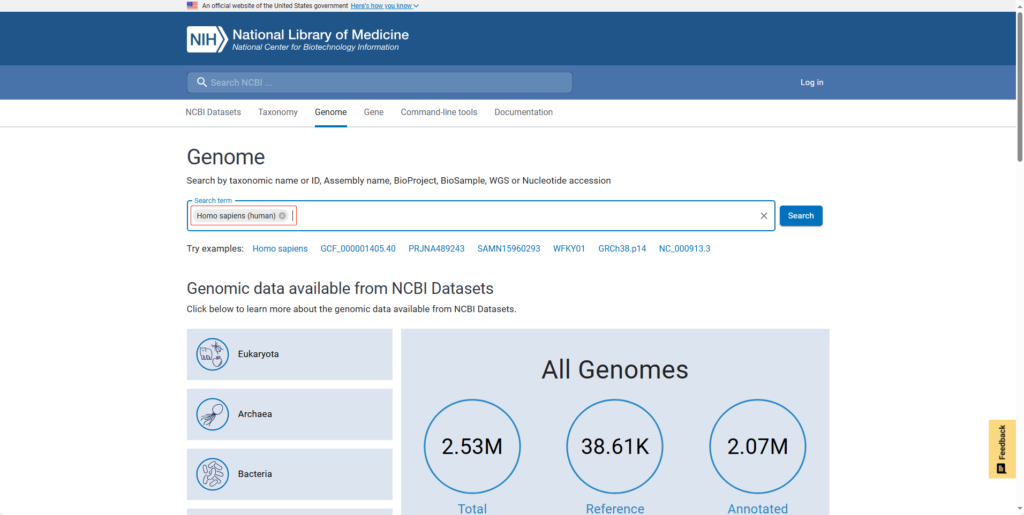
在弹出的assembly组装结果中选择一个(除非有指定的目标,一般都选择最上面有绿标“reference genome” 的)
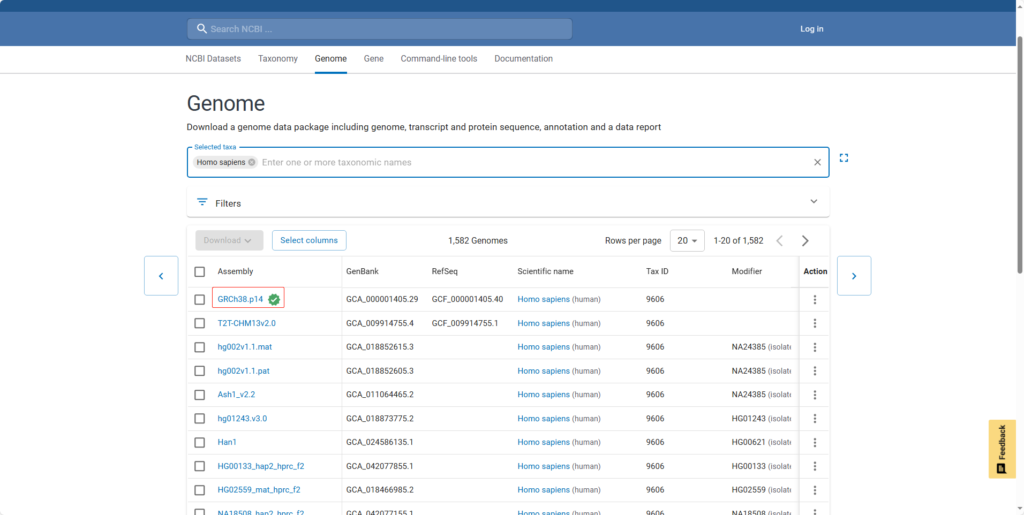
在这个界面可以看到基本介绍、组装统计、注释细节、质量分布、染色体等等基础信息。我们要下载的文件在FTP里面。
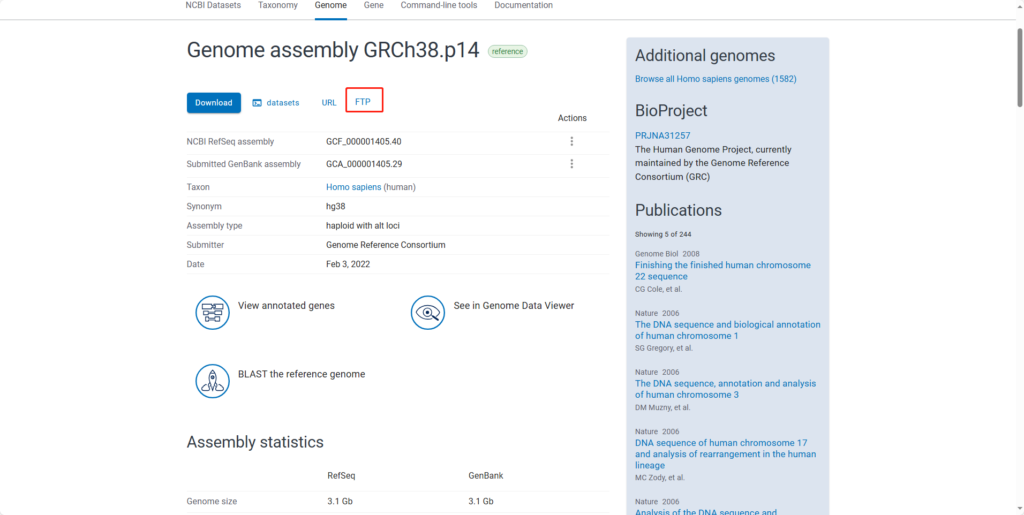
FPT下面即NCBI提供的各种用于下载的文本。一般选择.fna的参考基因组文件和.gtf的注释文件。(不同的物种在这个界面有很多不同的文件,有全基因组的、RNA部分的、CDS部分的……要根据需求选择。一般都选择genomic标签的,是最全的基因组。)
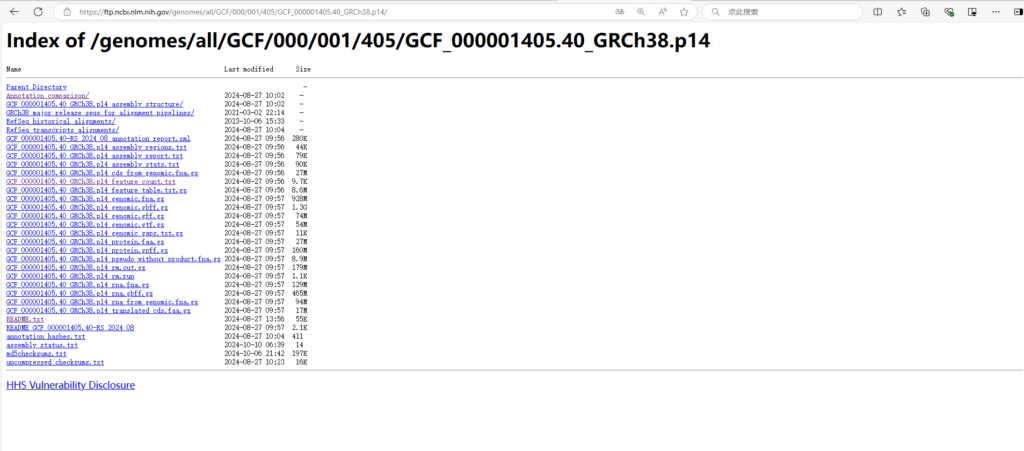
Tips:在NCBI有RefSeq、GenBank两个基因组数据库。
GenBank是一个开放的数据库,任何研究者或机构都可以提交序列数据,因此它包含了大量的序列信息,可能存在冗余和不准确的数据。
RefSeq数据库则是NCBI从GenBank和其他来源筛选出的非冗余序列集合,旨在提供更准确和可靠的参考序列。
从查询号即可看出区别:GCF开头是RefSeq、GCA开头是GenBank。(F :reference sequences;A :Assembly)
查询号第一次提交版本默认为1,后续作者提交更新版本,会在末尾加版本号 .2
Ensembl
从Ensembl下载参考基因组和注释信息:
在选择框中选择目标物种
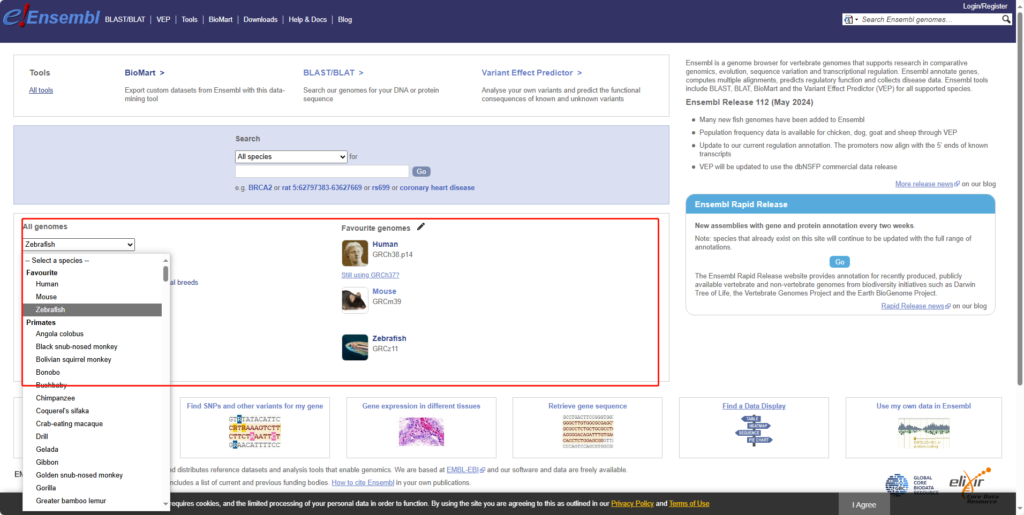
会转跳新页面,会有此物种的相关模块:Genome assembly、Gene annotation、Variation、Comparative genomics…
参考基因组从Genome assembly模块中的“Download DNA sequence ”下载;
注释文件从Gene annotation模块中的“Download GTF”下载。
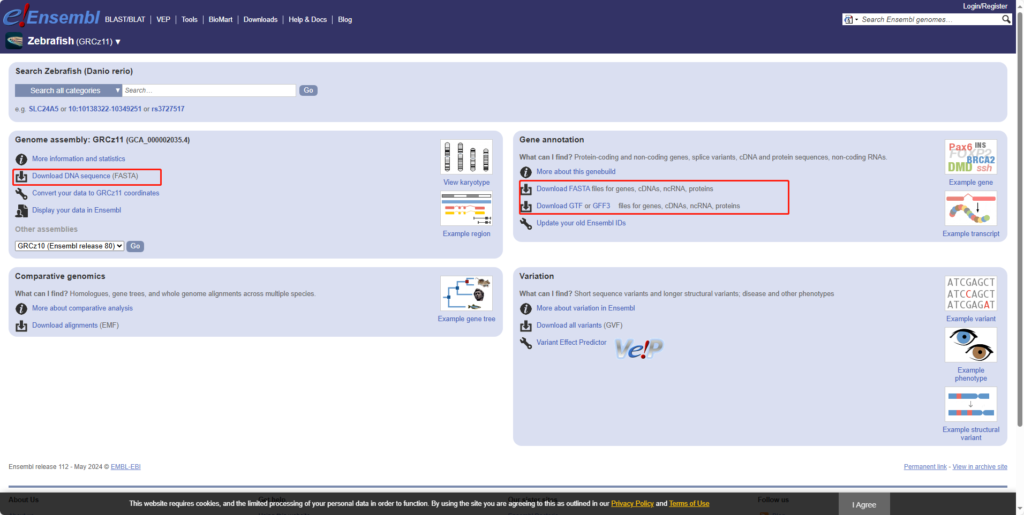
ensembl数据库的文件路径结构导致参考基因组和注释信息不在同一个子目录下,所以使用时需要注意。
首先ensembl数据库有一个release版本,这个是官方会不定期的进行大更新的网站版本。
在release版本下面再分为fasta、gff3、gtf、bed、genebank……等文件夹用于存放对应数据。在这些文件夹下即各种物种分类。
在fasta文件夹下面存放的是dna、cds、cdna、ncrna….等分类的fasta数据。
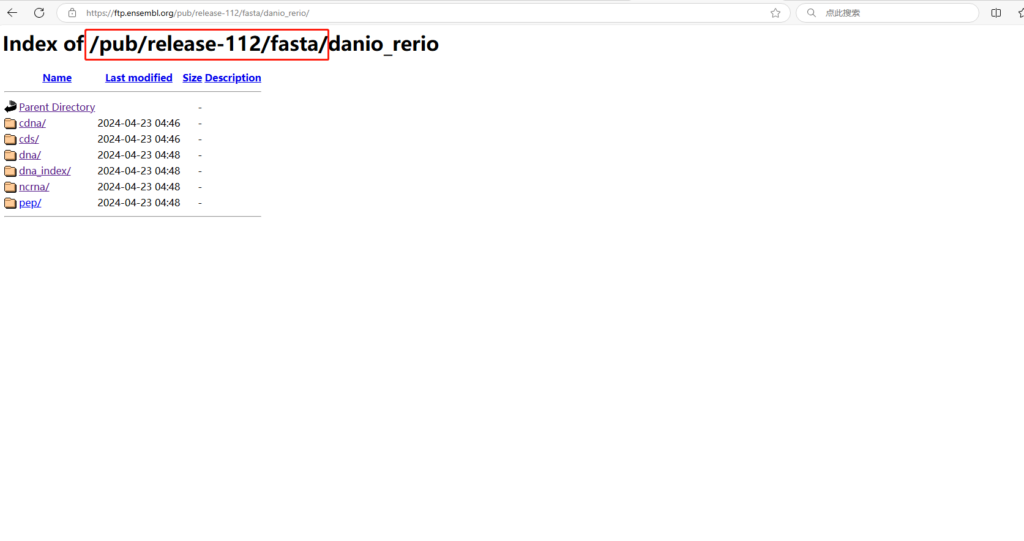
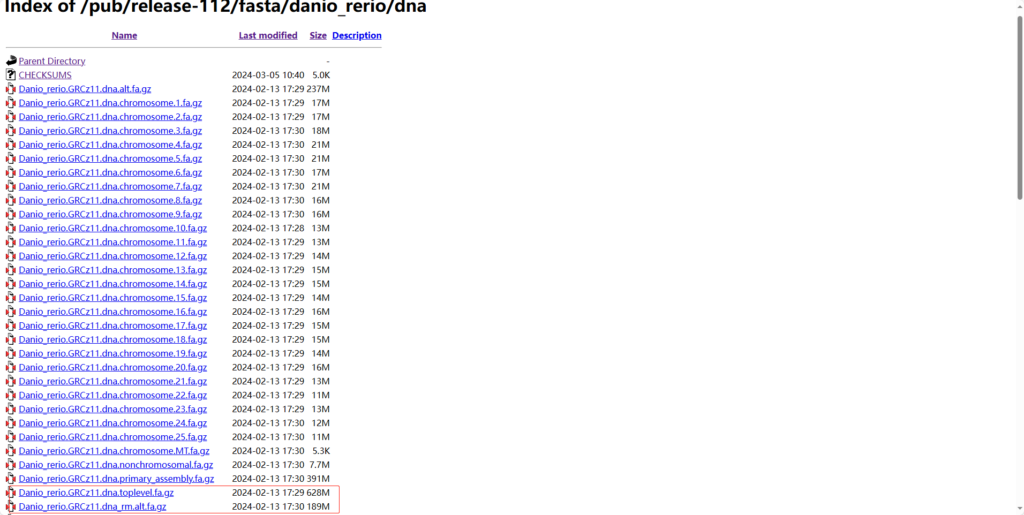
Tips:Ensembl数据库里的fasta文件有很多很多,他提供了各种非常细致的分类结果。
Ensemble提供两种组装形式和3种重复序列处理方式的参考基因组,分别是primary、toplevel 、unmasked(dna) 、soft-masked(dna_sm) 和masked(dna_rm) 。
dna、dna_rm和dna_sm三种类型
- ‘dna’ – unmasked genomic DNA sequences. 未处理的基因组序列
- ‘dna_rm’ – masked genomic DNA. 用“RepeatMasker tool”工具识别处理的基因组序列,重复区和低复杂区碱基用N替代
- ‘dna_sm’ – soft-masked genomic DNA. 重复区和低复杂区碱基用小写字母替代
primary、toplevel两种组装形式
- toplevel:包含所有染色体序列、未组装到染色体序列和用N填充的单倍型/补丁区域
- primary_assembly:用于序列比对的最完善的基因组,去除了单倍型/补丁区域
Masked基因组:指所有重复区和低复杂区被N代替的基因组序列, 比对时就不会有reads比对到这些区域。一般不推荐用masked的基因组, 因为它造成了信息的丢失, 由此带来的一个问题是uniquely比对到masked基因组上的reads实际上可能不是unique的。而且masked基因组还会带来比对错误, 使得在允许错配的情况下, 本来来自重复区的reads比对到基因组的其它位置。另外检测重复区和低复杂区的软件不可能是完美的,这就造成遮盖住的重复序列和低复杂区并不一定是100%准确和敏感的。
soft-masked基因组:是指把所有重复区和低复杂区的序列用小写字母标出的基因组,由于主要的比对软件,比如BWA、bowtie2等都忽略这些soft-mask,直接把小写字母当做大写字母比对, 所以使用soft-masked基因组的比对效果和使用unmasked基因组的比对效果是相同的。
因此,我们一般优先选择unmasked(dna) +primary_assembly 的组合。(如果没有primary_assembly,则选择toplevel 代替)
UCSC
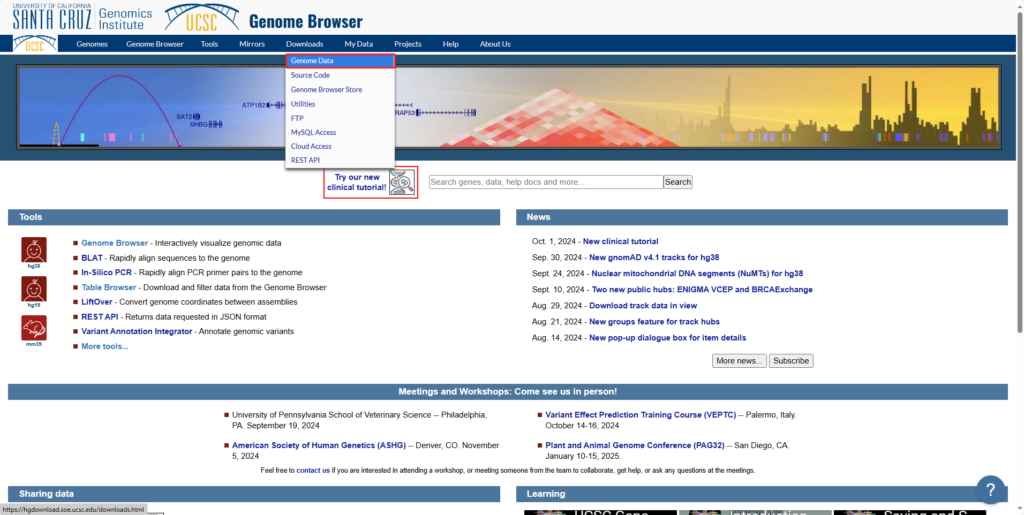
从UCSC下载参考基因组和注释信息:
在Genome Data中选择需要的物种
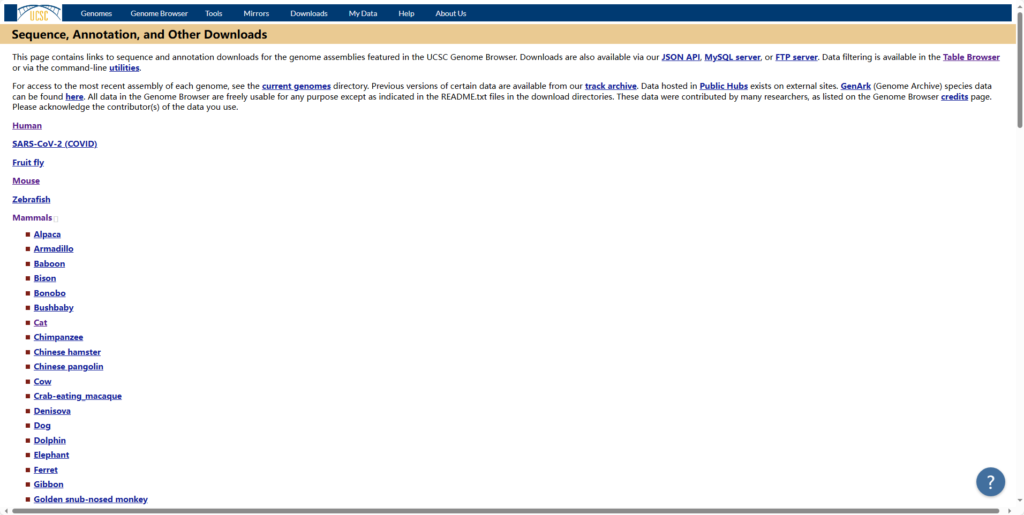
从物种里选择一个版本(一般都选择最新的)
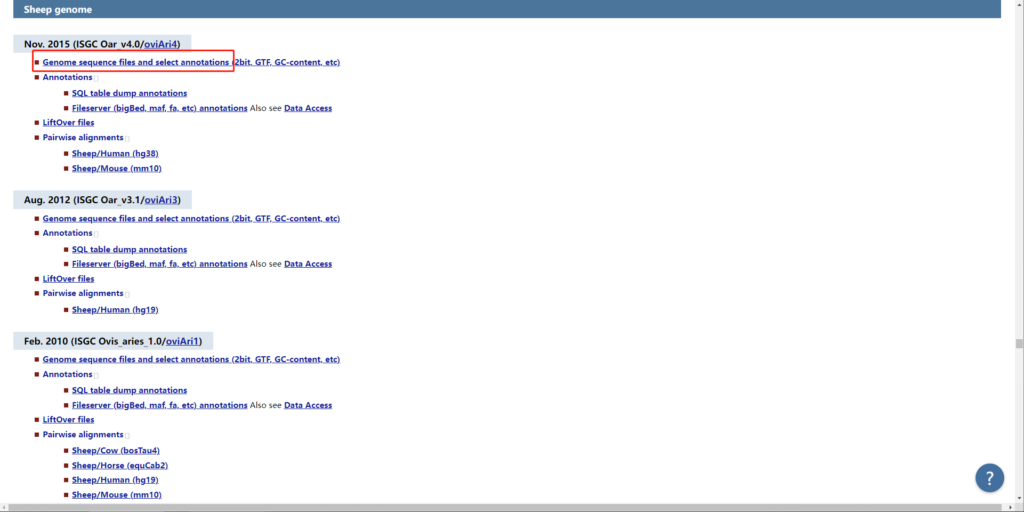
参考基因组和注释文件一般都是在“Genome sequence files and select annotations”目录下,
选择 “物种名.fa.gz” 的参考基因组;gtf文件在 genes/ 文件夹下。
GENCODE
Gencode只涉及小鼠和人两个物种,所以使用起来比较简单,只需要选择最新版本(current release)即可。
Gencode官网按照GTF / GFF3 files、Fasta files、Metadata files分为三类,分别都附带了详细的说明,按照自己的需求下载即可。
也可以使用FTP site下载FTP格式。
