DNA的转录、翻译是一个动态的过程,本文各个名词的解释开始介绍。介绍整个过程中遗传信息的传递,不同阶段产物的定义,名词解释。
基因包含编码区和非编码区:
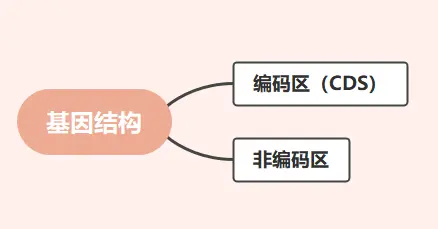
- 编码区:真核生物基因的编码区是不连续的,分为外显子和内含子,它们交替出现。在原核生物中,基因是连续的,也就是说无外显子和内含子之分。
- 非编码区:不能编码蛋白质的区段叫做非编码区。非编码区位于编码区前后,同属于一个基因,控制基因的表达和强弱 。
所以上图对于原核生物来说是正确的,但对于真核生物来说则不太详细了。
本篇将基于真核生物来进行介绍,原核生物的基因结构比较简单,本篇不介绍。

我们知道,基因DNA分为:编码区(Coding region),非编码区(Non-Coding region)。
编码区是指能够转录信使RNA(mRNA)的部分,它能够合成相应的蛋白质;而非编码区是不能够转录信使RNA的DNA结构,但是它能够调控遗传信息的表达,如启动子、终止子 分别位于编码区的上游和下游,负责调控基因的转录。
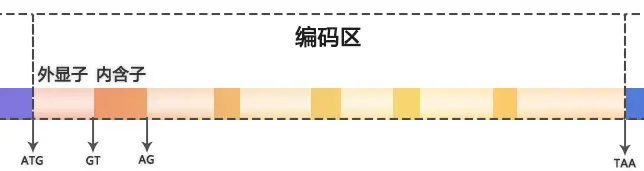
编码区的DNA转录出来hnRNA(mRNA的前体)包括了内含子(Intron)区域、外显子(Exon)区域。
外显子(expressed region)是真核生物基因的一部分。外显子是最后出现在成熟RNA中的基因序列,又称表达序列。 编码区中不连续的具有蛋白编码功能的DNA序列。
内含子(Intron)又称间隔顺序,指一个基因或mRNA分子中无编码作用的片段。就是编码区中外显子之间间隔的非编码序列。内含子在后续的生物过程终会被切掉。
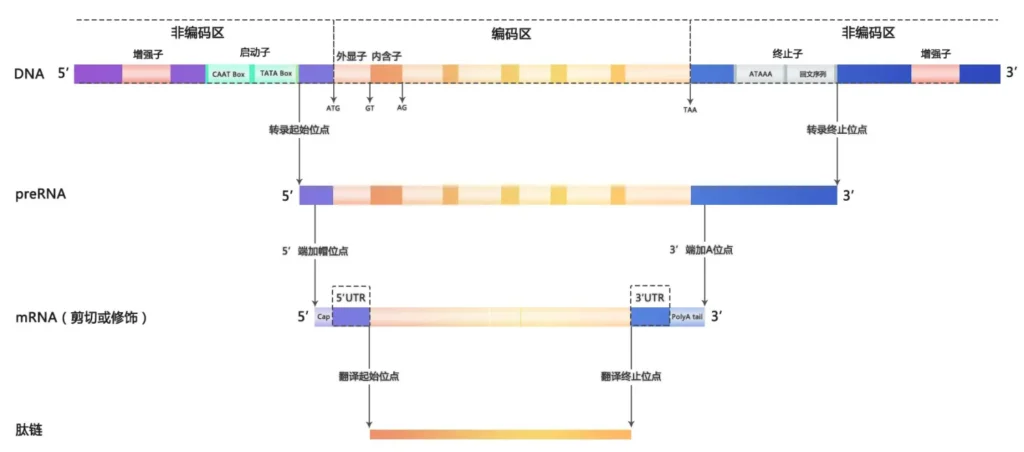
hnRNA经过一系列的剪切修饰总重形成的 成熟mRNA 中只包含外显子部分,此时的外显子序列中还包含了UTR区域和CDS区域。
UTR(UntranslatedRegions):mRNA两端的非编码序列
5’UTR:上游非编码区,位于5’帽子与起始密码子(AUG)之间,不包括起始密码子。
3’UTR:下游非编码区,位于编码区末端的终止密码子到3’尾巴之间,包含终止密码子。
CDS(Coding sequence):是指编码一段蛋白产物的序列,是与蛋白质密码子一一对应的DNA序列。本例中是 成熟的外显子序列中从起始密码子开始(包含起始密码子),到终止密码子为止的序列(不包含终止密码子)。
另外还有两个部分只做了解即可:
5’帽子:作用就是帮助mRNA跨过核膜,进入胞质,并且此过程中保护5‘不被降解;翻译时保证IFiii和核糖体识别
3’poly-A尾巴:作用也是帮助mRNA跨过核膜,进入胞质,并且增加了mRNA在胞质中存在的稳定性。因为mRNA的降解过程是随着时间延长,A尾逐渐变短;
另外还有一个重要的名词需要理解
开放阅读框(ORF:open reading frame):从起始密码子到终止密码子的碱基序列,且不包含终止密码子
编码区CDS(coding sequence):是指编码一段蛋白产物的序列,是与蛋白质密码子一一对应的DNA序列。
开放阅读框(Open reading frame):是指从一个起始密码子开始到一个终止密码子结束的一段序列。
※ CDS与ORF的联系与区别:
并不是所有读码框都能表达出蛋白产物,或者能表达出占有优势或者能产生生物学功能的蛋白;CDS必定是一个ORF,但也可能包括多个ORF,相反,每个ORF不一定都是CDS。
ORF: — translation(理论上存在的), CDS: — transcription(事实存在的)
因为内含子剪切等各种原因,ORF(理论上的)和CDS(实际存在的)不是一一对应关系。
